一、核心技术引领突破:CUT&Tag 技术与结核病研究新视角
在基因组学与表观遗传学研究领域,CUT&Tag(Cleavage Under Targets and Tagmentation) 技术正以其高灵敏度、低背景噪音和高效性,逐步革新我们对染色质结构与功能关系的认知。该技术的核心在于利用蛋白 A/Tn5 转座酶融合蛋白(pA-Tn5),在特异性抗体的引导下,实现对目标基因组区域的精准定位与标签化。
近日,一项发表于《Nature Communications》的重要研究,首次将 CUT&Tag 技术成功应用于原核生物——结核分枝杆菌(Mycobacterium tuberculosis, Mtb),系统揭示了其在氧化应激条件下独特的 G-四链体(G-quadruplex, G4)景观,文章题目为《CUT&Tag reveals unconventional G-quadruplex landscape in Mycobacterium tuberculosis in response to oxidative stress》。
结核病作为全球最致命的传染病之一,因其日益严峻的耐药性问题,亟需开发新型治疗靶点。G-四链体是一种由鸟嘌呤富集序列形成的核酸的替代二级结构,在真核生物中已被证实参与转录调控、基因组稳定性维持等多个关键生物学过程。结核分枝杆菌基因组具有异常高的 GC 含量(约 65.6%),理论上存在大量潜在的 G4 形成序列,然而这些结构是否在细菌体内真实存在、其分布规律及生物学功能,长期以来缺乏直接的实验证据。
本研究通过优化并应用 CUT&Tag 这一前沿技术,成功绘制了 Mtb 在标准生长条件及模拟宿主巨噬细胞内环境的氧化应激条件下的全基因组 G4 图谱。研究不仅回答了「G4 结构是否在 Mtb 体内形成」这一基础问题,更深入探讨了其在细菌适应宿主环境、响应氧化应激过程中的潜在作用,为针对结核分枝杆菌的新型抗菌策略开发开辟了全新道路。
二、CUT&Tag 技术的核心优势:为何选择它?
相较于传统的染色质免疫沉淀测序技术(ChIP-seq),CUT&Tag 在多个维度展现出颠覆性优势,这使其成为本次突破性研究的核心技术选择。
1. 操作流程更为简捷,且对细胞状态干扰更小
该技术无需进行甲醛交联和超声片段化这两个在 ChIP-seq 中至关重要的步骤。CUT&Tag 直接在透化的完整细胞核内进行反应,由抗体引导的 pA-Tn5 转座酶在目标位点原位进行 DNA 切割并同时添加测序接头,更好地保留了体内真实的生物学状态。
2. 具有卓越的信噪比和极低的起始材料要求
本研究指出,利用 CUT&Tag 技术仅需约 5 百万条 reads 即可获得高质量的 RNA 聚合酶定位数据,而传统的 ChIP-seq 技术通常需要至少五倍以上的测序深度。这种高效率源于其极低的背景噪音,因为 Tn5 转座酶的活性被严格限制在抗体结合的特定区域。在研究初期,作者尝试使用 ChIP-seq 定位 G4 结构,但遇到了背景信号过高、难以准确识别真实峰值的挑战;而转为 CUT&Tag 后,信噪比得到显著改善,获得了清晰可靠的 G4 定位结果。
3. CUT&Tag 技术重现性高,且易于实现多组学整合
本研究中的生物学重复表现出高度的相关性,确保了数据的可靠性。同时,该技术能够轻松地与 RNA-seq 等转录组数据相结合,从而直接关联特定染色质特征(如 G4 形成)与基因表达变化,为机制阐释提供了强有力的支持。
三、关键图表解析:实验发现与核心结论
技术奠基:优化 CUT&Tag 流程并验证其在分枝杆菌中的适用性
为确保 CUT&Tag 技术在结核分枝杆菌这一复杂原核系统中的可靠性,研究团队首先针对 RNA 聚合酶 β 亚基进行了全基因组定位分析,因其已有可靠的 ChIP-seq 数据可供对比。实验流程中,研究人员对细菌细胞进行了温和的甲醛固定,随后利用透化处理使抗体和 pA-Tn5 转座酶能够接近并结合其基因组目标位点。经过优化的标签化反应后,通过 PCR 扩增构建测序文库。每个实验条件均设置了三个生物学重复,并采用 IgG 作为阴性对照,以确保结果的特异性。
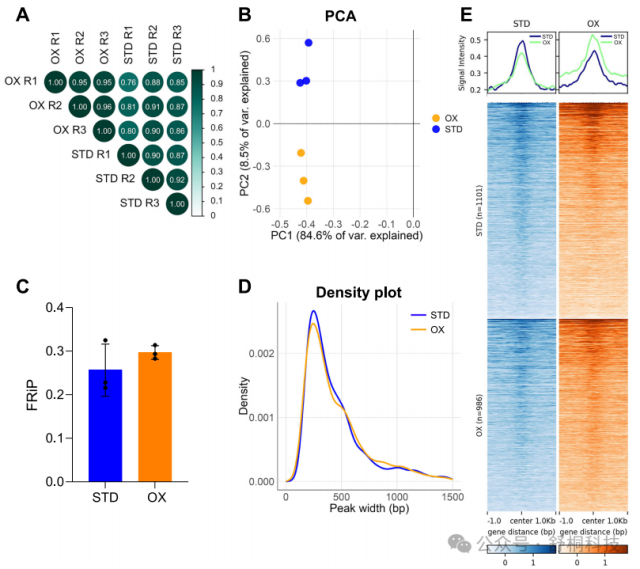
图 1. RNA Pol-CUT&Tag 在 Mtb 基因组中的验证
重要实验结论:
1. 针对 RNA 聚合酶的 CUT&Tag 实验在标准培养和氧化应激条件下均显示出高度的重复性,生物学重复间具有极高的 Pearson 相关系数,表明重复性良好,证明了该方法在 Mtb 中的稳定性和可靠性。(图 1A)
2. 主成分分析清晰地将标准培养和氧化应激条件下的样本区分开,表明 CUT&Tag 数据能够敏感地捕捉由环境变化引起的生物学差异。(图 1B)
3. 所有样本达到了适当的测序深度,FRIP 值均远超 0.01 的成功实验阈值,表明测序数据中位于峰值区域的 reads 比例很高,数据质量优异。(图 1C)
4. 鉴定出的峰值大小分布中位数约为 200 bp,符合 CUT&Tag 技术的预期特征。(图 1D)
5. 在高置信度峰值中心区域,信号密度热图显示出强烈的、一致的富集模式,直观地证实了免疫沉淀和目标定位的效率。(图 1E)
转录蓝图:揭示 Mtb 在不同应激条件下的转录活性景观
利用已验证的 CUT&Tag 流程,研究人员系统性地描绘了 RNA 聚合酶在 Mtb 全基因组的结合位点。在标准生长条件下鉴定出 1101 个高置信度峰值,而在氧化应激条件下鉴定出 986 个。通过对峰值进行基因组特征注释,并结合已发表的 RNA-seq 数据,他们深入分析了 RNA 聚合酶结合与基因表达水平之间的关联。
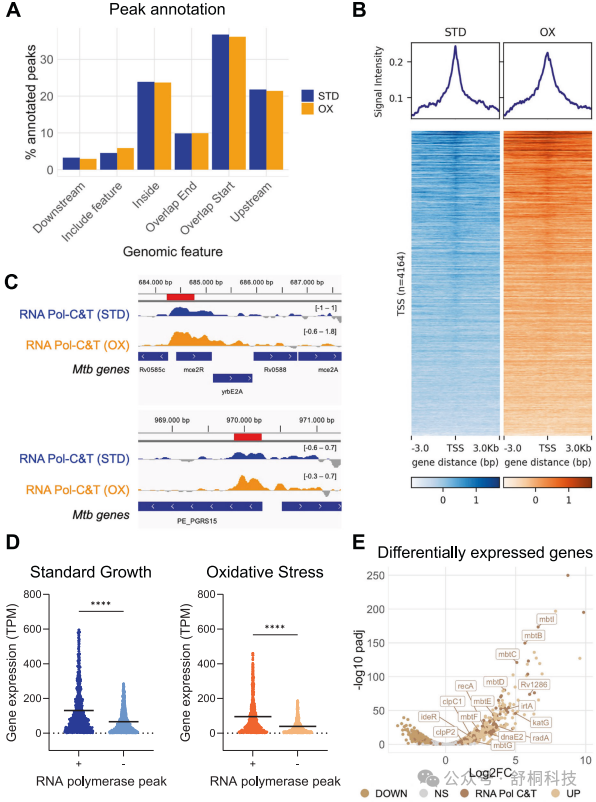
图 2. 通过 CUT&Tag 在 Mtb 中进行全基因组 RNA 聚合酶分析
重要实验结论:
1. RNA 聚合酶峰值主要富集在基因转录起始位点附近,在标准和氧化应激条件下分别占 36.7% 和 36.1%,这与 RNA 聚合酶准备启动转录的功能相符。(图 2A)
2. 有相当比例的峰值位于基因内部和基因上游区域,揭示了 RNA 聚合酶在整个转录周期中的动态分布。(图 2A)
3. 在转录起始位点两侧的信号密度热图显示 RNA 聚合酶信号在 TSS 处达到峰值,进一步印证了其在转录起始中的核心作用。(图 2B)
4. 已知的 RNA 聚合酶结合位点,如 rv0586 和 rv0872c,在本研究的 CUT&Tag 数据中同样被检测到,与既往研究相互印证,验证了数据的准确性。(图 2C)
5. 携带 RNA 聚合酶 CUT&Tag 峰值的基因,其表达水平显著高于没有峰值的基因,这直接证明了 RNA 聚合酶结合与活跃转录之间的正相关关系。(图 2D)
6. 在氧化应激条件下,大量差异表达基因与 RNA 聚合酶结合变化相关,其中包括多个已知的应激反应基因,如过氧化氢酶基因 katG,表明 CUT&Tag 能有效捕捉应激响应中的转录重编程。(图 2E)
结构异类:揭示结核菌内以双链 tetrad 为主的基因体内 G-四链体
研究团队随后应用 BG4 抗体进行 G4-CUT&Tag 实验,旨在绘制 Mtb 基因组中的 G4 结构图谱。实验采用了与研究 RNA 聚合酶时相似的 CUT&Tag 流程,但使用了针对 G4 结构的 BG4 抗体。通过对免疫沉淀序列进行基序分析、G4 形成潜能预测以及圆二色光谱验证,全面表征了 Mtb G4 结构的序列特征和折叠特性。
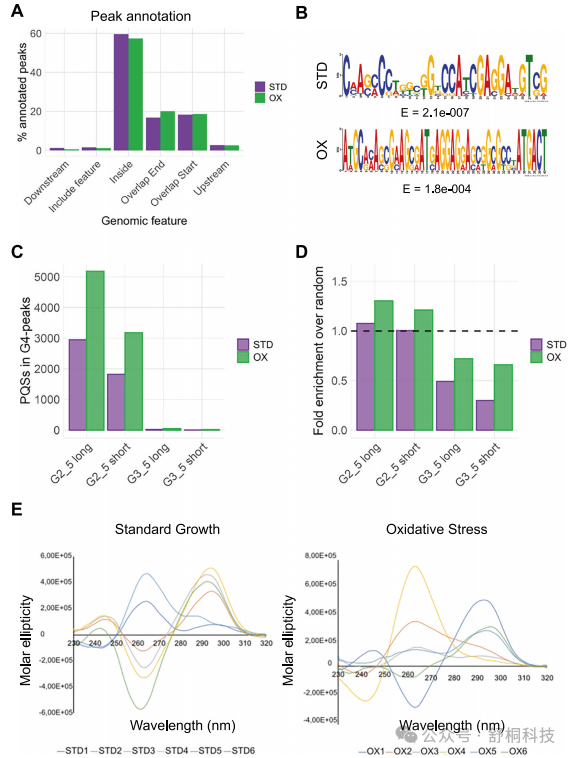
图 3. Mtb 基因组中的 G4-CUT&Tag
重要实验结论:
1. G4 结构在 Mtb 中的分布与真核生物截然不同,近 60% 的 G4 峰值位于基因编码区内,而位于启动子区域的不足 3%,提示其在细菌中可能扮演不同的调控角色。(图 3A)
2. 从 G4 峰值序列中鉴定出的富 G 基序,证实了这些区域确实具备形成 G4 结构的序列基础。(图 3B)
3. 约 99% 的预测 G4 形成序列由双鸟嘌呤链和短环组成,而非经典 G4 所要求的三束或四束,这通常预示着结构稳定性较低但动态性更强。(图 3C)
4. 随机化统计分析表明,双 tetrad G4 序列在实验数据中的富集具有统计学显著性,而非随机出现;同时,三分体(3-tetrad)G4s 的显著缺失也证明了该发现的特异性。(图 3D)
5. 圆二色光谱分析证实,从 CUT&Tag 实验中回收的多个代表性序列在体外确实能够折叠成 G4 结构,大多呈现反平行拓扑构象,为体内 G4 的形成提供了直接的生化证据。(图 3E)
应激重塑:氧化压力特异性诱导 G4 形成并关联基因表达抑制
为了探究环境压力如何影响 G4 结构的形成,研究人员对标准生长和氧化应激条件下的 G4-CUT&Tag 数据进行了全面的比对分析。他们不仅关注 G4 峰值数量的全局变化,更通过严格的统计学方法鉴定出条件特异性的 G4 位点,并将这些表观遗传信息与 RNA-seq 获得的转录组数据深度整合,以揭示 G4 动态变化的功能性后果。
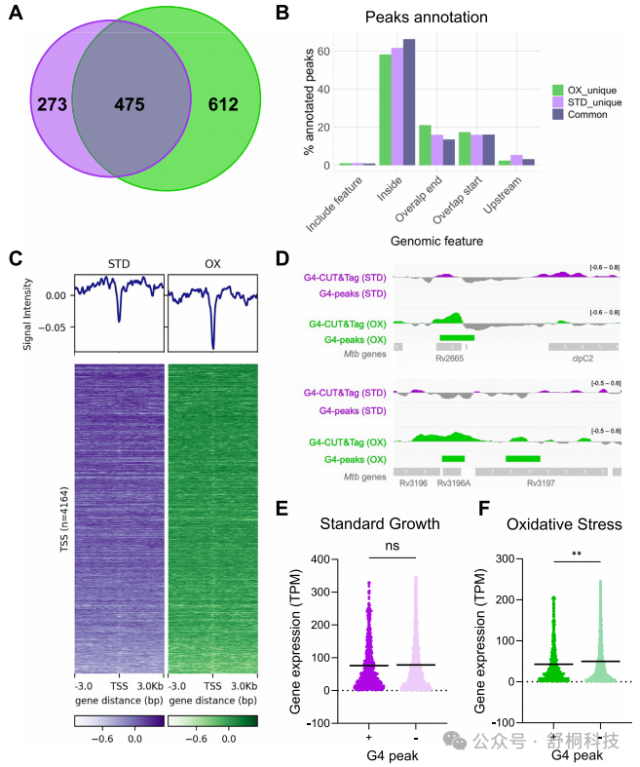
图 4. 氧化应激条件与标准生长条件相比的 G4 富集
重要实验结论:
1. 氧化应激是 G4 形成的强力诱导因素:在氧化应激条件下,研究人员鉴定出 1087 个高置信度 G4 峰值,数量显著多于标准生长条件下的 748 个。更为关键的是,超过 50% 的 G4 峰值为氧化应激条件所特有,而标准条件特有的峰值占比不足 40%,这清晰地表明氧化应激能有效诱导体内 G4 结构的重新形成与特定位点的稳定。(图 4A)
2. Mtb 的 G4 始终偏好基因体内分布:无论 G4 峰值是两种条件共有,还是某一条件所特有,其基因组分布都高度一致地集中在基因编码区内,占比均接近 60%。这一发现有力地维持了 Mtb G4 结构「非典型分布」的核心特征,强调了其与真核模式的根本差异。(图 4B)
3. G4 在启动子区域普遍缺失:对基因转录起始位点上下游的信号分析显示,G4-CUT&Tag 信号并未在 TSS 区域出现富集峰。这与真核生物中 G4 通常富集于核小体缺失的启动子区以调控转录起始的经典模式形成了鲜明对比。(图 4C)
4. G4 与基因表达的关联具有条件依赖性:在正常生长状态下,基因组中 G4 的存在与基因的转录水平之间并未发现显著的相关性,暗示此时 G4 可能不直接参与广泛的转录调控。(图 4E)
5. 氧化应激下 G4 成为转录抑制标志:然而,在氧化应激条件下,携带 G4 结构的基因,其平均转录水平显著低于不携带 G4 的基因。这一强烈的负相关关系提示,在压力环境下,G4 的形成与基因表达的主动下调存在着密切的功能关联。(图 4F)
功能关联:特异性 G4 直接参与转录抑制并调控关键适应通路
在发现氧化应激能诱导大量特异性 G4 形成并与基因下调相关后,研究人员进一步深入分析了这些特异性 G4 的调控机制与生物学功能。他们精细地分析了 RNA 聚合酶在这些位点的占用情况,并将基因按表达水平分层,以剖析 G4 在不同转录活跃度背景下的影响,最后通过生物信息学方法揭示了这些 G4 相关基因的共同功能。
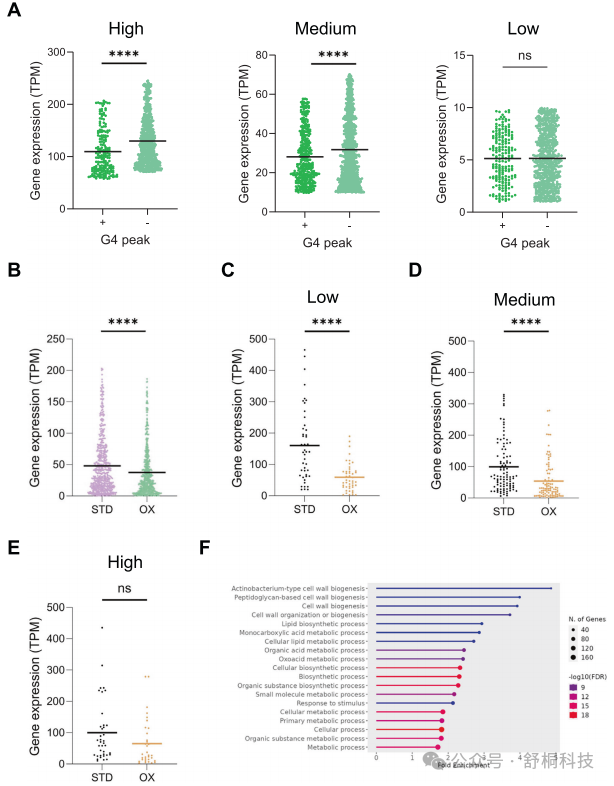
图 5. 仅在氧化应激条件下出现 G4 结构的基因
重要实验结论:
1. G4 的抑制效应在活跃转录基因中最为显著:当把基因按其表达水平分为高、中、低三组后,发现在氧化应激条件下,G4 对于高表达和中表达基因的转录抑制效应最为强烈和显著,而对于本就低表达的基因则影响甚微。这表明 G4 可能主要作为高活性转录基因的「分子刹车」。(图 5A)
2. 证实 G4 形成与基因下调的因果关系:通过追踪那些「仅在氧化应激下形成 G4」的基因,研究发现这些基因在应激条件下的表达水平,相比它们在正常条件下的水平出现了显著下降。这一纵向比较为「G4 诱导直接导致转录抑制」提供了强有力的因果证据。(图 5B)
3. G4 通过阻碍转录延伸发挥作用:通过分析 RNA 聚合酶在 G4 位点的占用变化,研究发现,即便在 RNA 聚合酶结合并未减少(甚至有所增加)的情况下,G4 的存在依然与转录效率的降低相关。这强烈暗示,G4 结构并非通过阻止转录起始,而是通过直接阻碍转录延伸过程来发挥抑制功能。(图 5C, D, E)
4. G4 相关基因富集于关键适应通路:基因本体富集分析显示,这些氧化应激特异性 G4 所关联的基因,并非随机分布,而是显著富集在「代谢过程」、「生物合成过程」及「细胞壁组织与生物发生」等关键功能类别。这些通路的下调,恰好与结核分枝杆菌在宿主压力环境下为求生存而主动减缓代谢、进入休眠适应状态的经典策略高度一致。(图 5F)
四、总结
本研究首次将 CUT&Tag 技术成功应用于原核生物系统,完成了结核分枝杆菌中转录机器和 G4 结构的高分辨率定位。研究发现颠覆了我们对细菌中 G4 结构的传统认知:它们主要分布于基因内部而非启动子区,且主要由非经典的双鸟嘌呤束构成。更重要的是,研究揭示了氧化应激能显著诱导 G4 结构的形成,并且这些结构与基因表达的抑制密切相关,提示 G4 可能在 Mtb 应对宿主免疫压力、调控适应性反应中扮演了关键「分子开关」的角色。这些发现不仅深化了我们对细菌表观遗传调控的理解,更为开发以 G4 为靶点的新型抗结核药物奠定了坚实的理论基础。
本研究的成功,高度依赖于 CUT&Tag 技术的核心组件——Tn5 转座酶及其复合体的性能。其中,pA-Tn5 转座酶/转座体是实现抗体导向性标签化的关键,其活性和特异性直接决定了实验的信噪比和成功率。
五、舒桐科技解决方案
舒桐科技深刻理解前沿技术对核心试剂的苛刻要求,依托强大的自主研发平台,推出了一系列高性能的 Tn5 转座酶及相关产品,为您的 CUT&Tag 研究提供全面、可靠的解决方案:
1. pA-Tn5 转座酶 & pA-Tn5 转座体:蛋白 A 与高活性 Tn5 酶的完美融合,适用于绝大多数 IgG 类型抗体,确保高效、特异的靶向标签化。
2. pG-Tn5 转座酶 & pG-Tn5 转座体:蛋白 G 与 Tn5 酶的融合产品,对小鼠 IgG1 等特定抗体亚型具有更强的结合力,为您提供更灵活的抗体选择。
3. pA/G-Tn5 转座体:融合了蛋白 A 和蛋白 G 的双重结合域,能够高效结合几乎所有种类的 IgG 抗体,包括兔、鼠、羊、牛、人等多种来源的抗体及其各种亚型,特别是对难以结合的小鼠 IgG1 表现出优异的亲和力,是进行多样化研究设计的理想选择。
此外,舒桐科技还提供与 CUT&Tag 技术完美配套的高效建库试剂盒。该试剂盒经过专门优化,可与上述 Tn5 转座体无缝衔接,简化实验操作流程,更好得提升文库构建的效率和质量,确保您能获得高质量的测序数据。
对于希望快速开展研究或需专业支持的客户,舒桐科技更提供一站式的 「CUT&Tag 测序及分析」服务,包括样本处理、文库构建、高通量测序以及专业的生物信息学分析。
对于希望进行多组学分析的客户,我们也提供专业的整合分析解决方案。通过将 CUT&Tag 测序与 RNA-seq 联合分析,我们的专业团队能够帮助您构建从表观遗传调控到基因表达的完整调控网络。
最后,舒桐也提供定制化的分析服务,确保您能精准、高效地获得并解读基因组学数据,加速您的科研发现。
从核心酶原料到完整的检测服务,舒桐科技致力于成为您创新科研之路上最值得信赖的合作伙伴。我们期待与您携手,共同探索生命科学的未知领域,推动研究成果的转化与应用。