Takara 为了满足广大研究人员的需要提供各种 PCR 酶。其中 PrimeSTAR®系列以其高保真性和高扩增效率作为「便于使用的高保真酶」受到广大研究人员的好评。在本稿中介绍实际进行的重复序列扩增例及在长链目的基因的特异性位点进行的突变导入例,验证 PrimeSTAR®系列的保真性。
■ 实验例 1:与各种 PCR 酶的保真性比较(1)
~ 富含 GC 序列的目的基因~
本公司为了验证 PCR 酶的保真性,选择实际的方法对多个扩增产物测序后比较错配率。扩增的目的基因序列设置在容易导入基因突变的富含 GC 序列区域。
【方法】
以富含 GC 序列并容易发生碱基突变的 Thermus thermophiles HB8 基因组 DNA 为模板,任选 10 个区域(各扩增片段约为 500 bp),使用各种 PCR 酶进行 PCR 扩增(反应液组份及 PCR 反应条件使用各酶推荐的操作流程)。将各自 PCR 扩增产物克隆至载体中,并对每种序列挑取复数的克隆进行测序确认碱基序列。以错配碱基数对总解析碱基数的比率来测定 mutant frequency(突变率)。
【结果】
PrimeSTAR®系列各种 PCR 酶的扩增产物(约 50 万个碱基)的测序结果表明,仅 10~30 个碱基发生了错配。可以看出其保真性优于高保真酶的起源酶 Pfu DNA Polymerase(图 1)。
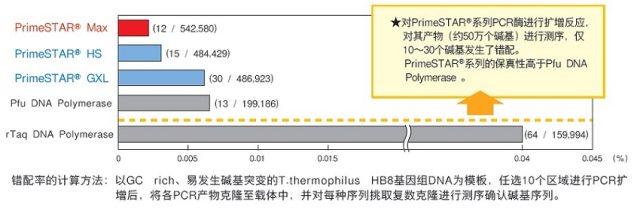
图 1. PrimeSTAR® 系列与各种 PCR 酶的保真性比较
■ 实验例 2:与各种 PCR 酶的保真性比较(2)
~ 重复序列~
本公司为了验证 PCR 酶的保真性,选择实际的方法对多个扩增产物测序后比较错配率。扩增的目的基因序列设置在容易导入基因突变的富含 GC 序列区域。
【方法 1】
含-(GA)8-或-(CA)8-重复序列的 500 bp DNA 片段(在 λDNA 来源的 DNA 序列中插入了重复序列)使用各种 PCR 酶,按照各酶的操作流程进行 PCR 扩增。将各自 PCR 扩增产物克隆至载体中,并对每种序列挑取复数的克隆进行测序确认碱基序列。以在重复序列中产生的碱基缺失及插入计算错配率。
【方法 2】
含-T26-重复序列的 500 bp DNA 片段(人基因组来源的 DNA 序列在质粒中克隆后的 DNA 片段)使用各种 PCR 酶进行 PCR 扩增、克隆、对复数的克隆测序后计数 T 碱基数。
【结果】
重复序列的复制错误是因为 PCR 酶对模板的识别能力下降,校正活性(3' → 5'exonuclease 活性)不能修复。因此即使是具有校正活性的高保真 α 型 DNA polymerase,与不具备校正活性的 Taq DNA polymerase 相比,其复制重复序列的保真性不一定更好。
从本次实验结果可以看出,添加独自研发的延伸因子的大幅提高了酶合成能力(processivity)的 PrimeSTAR® Max 和 PrimeSTAR® GXL,在扩增重复序列时具有优良的保真性(表 1、图 2)。
表 1. 各种 PCR 酶对重复序列 [-(GA)8-、-(CA)8-的保真性
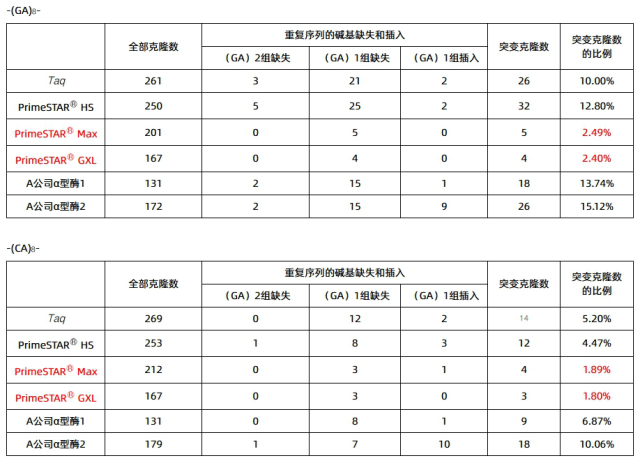
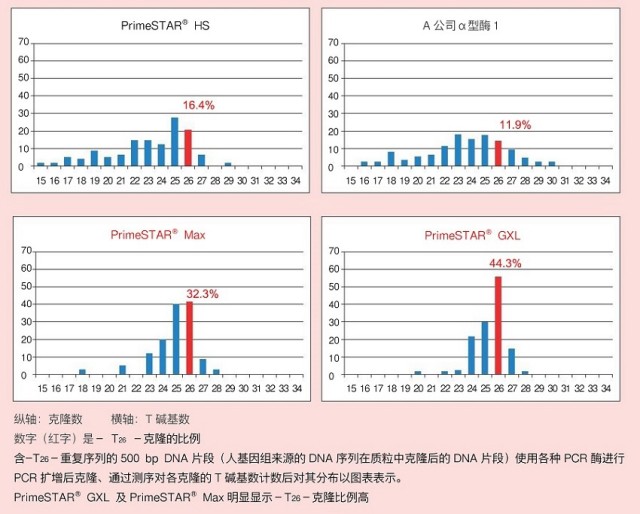
图 2. 各种 PCR 酶对重复序列 [-T26-] 保真性
更多产品信息可以联系我们:
Takara 预混型高保真酶 PrimeSTAR HS(Premix)
Takara 高保真酶 PrimeSTAR HS DNA Polymerase
Takara 高 GC 扩增用高保真酶 PrimeSTAR HS DNA Polymerase with GC Buffer