近日,领星与中国人民解放军总医院肿瘤学部焦顺昌教授团队合作撰写的学术论文「Predictive Biomarkers of Dicycloplatin Resistance or Susceptibility in Prostate Cancer」,发表于「Frontiers in genetics」,IF:4.599。中国人民解放军总医院副主任医师刘茗露博士为本研究论文的第一作者。本研究探究了前列腺癌双环铂耐药敏感生物标记物,为后续精准治疗提供分子证据。

研究背景(Background)
前列腺癌是男性群体中最常见恶性肿瘤,其死亡率位居第二位。使用激素治疗能够提升患者临床获益,但约有 10%-20% 的患者会出现耐药,且发展为去势抵抗前列腺癌。
铂类化疗药能够与细胞 DNA 交联抑制核酸复制和细胞分裂,进而达到抗肿瘤的作用。然而,前列腺癌患者接受铂类化疗药仅能延长 3-6 个月的总生存时间,且患者的副作用较大。
双环铂属于卡铂的衍生物,具有易溶解且更加稳定的特点。因此,双环铂被应用于一些肿瘤患者化疗方案中,如非小细胞肺癌、前列腺癌等。遗憾的是,仅部分患者对治疗有应答,因此探究有效预测双环铂疗效的生物标志物,能够提前筛选可能获益的患者。
WES 测序是一种高通量检测技术,其能够解析患者 DNA 序列以及基因拷贝数变异等基因信息。本项研究中,研究者共收集 16 例前列腺癌患者血液 cfDNA 及白细胞 DNA(对照),根据对双环铂应答率将患者分为「敏感度」和「耐药组」。根据两组 DNA 变异层面分析,探究可能与治疗疗效相关的有效生物标记物。
研究结果(Results)
1,患者基线数据分析
本研究共招募 16 例前列腺癌患者,其中 9 例对双环铂治疗敏感(6 PR + 3 CR),7 例对双环铂治疗不敏感(1 SD + 6 PD),所有患者的总治疗应答率为 56.3%。通过分析其他临床因素,如年龄、烟酒史、肿瘤级别及放化疗等,证实在两组患者之间均无显著差异。
(注:PR, partial response; CR, complete response; SD, stable disease; PD, progressive disease)
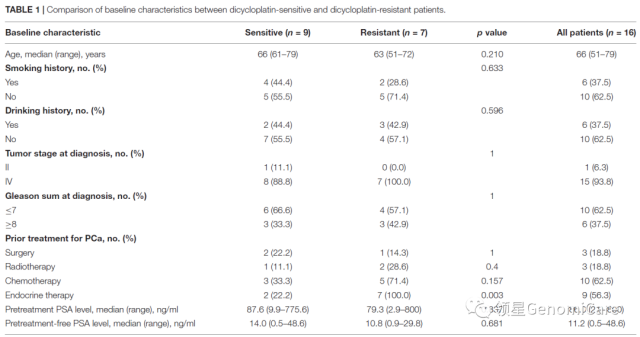
▲ 患者基线数据分析
2,TMB 及 MSI
首先根据「敏感组」与「耐药组」患者 WES 测序比对分析,获取每组患者 CNV 及 SNV 变异情况。
通过对两组患者 TMB 及 MSI 分析显示,「敏感组」和「对照组」患者的 TMB 和 MSI 均无显著差异,提示上述两个指标不能预测患者对双环铂的应答。
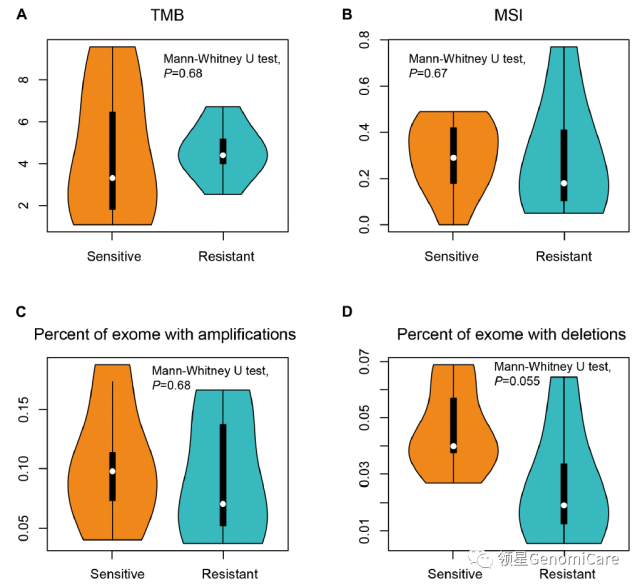
▲ 患者 TMB 及 MSI 分析
3,DNA 突变及拷贝数变异
16 例患者中最常见的体细胞突变基因为 ANKRD36C(56.3%)、KIAA2018(56.3%)、MUC4(56.3%)、TMBIM4(50.0%)和 RNF145(50.0%)。
通过分析「敏感组」和「耐药组」患者基因突变及拷贝数变异,鉴定出「耐药组」患者中明显高于「敏感组」的 7 个突变基因:SP8、HNRNPCL1、FRG1、RBM25、MUC16、ASTE1 和 TMBIM4(Fisher’s exact test, p < 0.05)。
通过分析患者携带的 CNVs 变异,提示 LAMTOR5-AS1、JUND 和 SCAND1 是常见的 CNV 缺失基因,且 CNV 缺失基因的数量大于 CNV 扩增基因的数量。CTAGE4、GAGE2E、GAGE2C 和 HORMAD1 基因拷贝数变异在「耐药组」患者中高于「敏感组」(Fisher’s exact test, p < 0.05),VARS、TMSB4Y、MUC22、DPCR1、CDSN 和 HISTCH2AC 基因拷贝数变异在「耐药组」患者中低于「敏感组」(Fisher’s exact test, p < 0.05)。
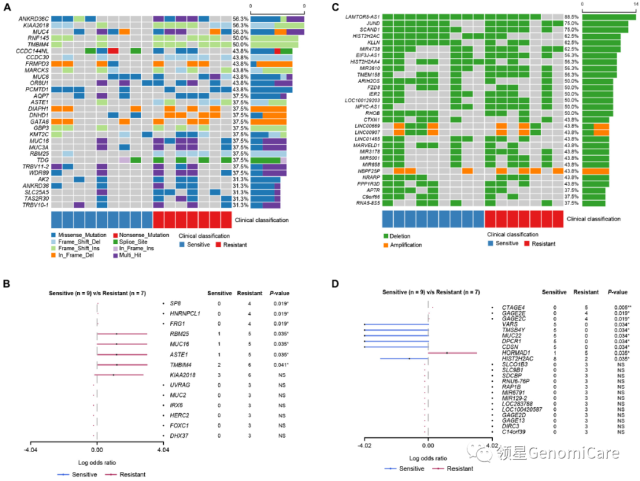
▲ 患者基因突变及拷贝数变异
4,两组患者突变类型
对各组患者突变碱基进一步分析发现,C>T 碱基突变是所有患者中最为常见的突变类型,而 T>G 突变在敏感组患者中较为常见。
基因 Signature3 和 Signature1 在所有患者中占较高的比例,分别为 40.1% 和 8.1%。Signature12 在「耐药组」患者中高于「敏感组」。
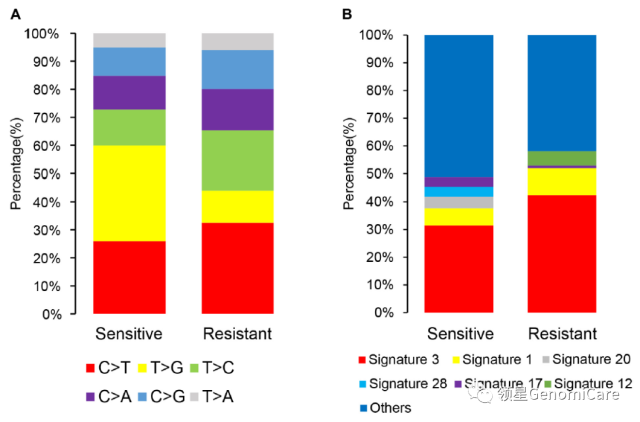
▲ 不同突变特征分析
5,单个基因预测疗效的敏感性与特异性
随后,分析两组患者差异突变基因和差异拷贝数变异与预测双环铂疗效的关系。其中,3 个突变基因(SP8、HNRNPCL1、FRG1)和 2 个 CNV 基因(GAGE2E、GAGE2C)的敏感性为 57.1%,特异性为 100%;其他 3 个突变基因(RBM25、MUC16 和 ASTE1)的敏感性和特异性分别为 71.4% 和 88.9%。
由于使用单一的基因作为预测疗效标记物,敏感性和特异性相对较低,这说明使用单基因无法准确地预测可能获益的患者。
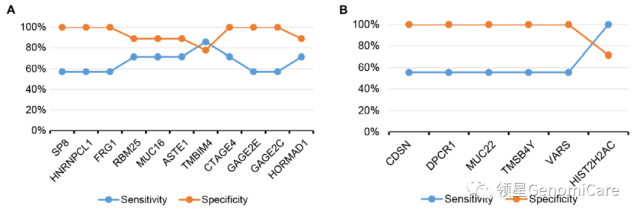
▲ 单个基因预测灵敏度和特异性
5,多基因组合预测生物标记物
最后,研究者考虑使用多基因组合的模型,增加预测双环铂疗效的敏感度和特异性。通过对多个突变基因和拷贝数变异基因重组分析发现,当同时使用 2 个突变基因(SP8/HNRNPCL1 或者 SP8/FRG1)和 1 个拷贝数缺失基因(GAGE2C)的预测组合时,预测的敏感度及特异性均能达到 100%。此外,使用 2 个拷贝数缺失基因(DPCR1 和 TMSB4Y)组合,灵敏度和特异性分别达到 88.9% 和 100%。

▲ 多基因预测灵敏度和特异性
研究结论(Conclusions)
本文通过对前列腺癌患者血液 cfDNA 进行外显子测序,分析了患者携带肿瘤突变基因。同时,根据患者对双环铂敏感性不同,筛选及提出了多基因组合模型,准确预测可能获益的患者,为后期精准治疗提供一定的分子依据。
参考文献(References):
1. Alexandrov, L. B., Jones, P. H., Wedge, D. C., Sale, J. E., Campbell, P. J., Nik-Zainal, S., et al. (2015). Clock-like mutational processes in human somatic cells. Nat. Genet. 47, 1402–1407.
2. Bos, M. K., Angus, L., Nasserinejad, K., Jager, A., Jansen, M. P. H. M., Martens, J. W. M., et al. (2020). Whole exome sequencing of cell-free DNA: A systematic review and Bayesian individual patient data meta-analysis. Cancer Treat. Rev. 83:101951.
3. Chang, W., Ma, L., Lin, L., Gu, L., Liu, X., Cai, H., et al. (2009). Identification of novel hub genes associated with liver metastasis of gastric cancer. Int. J. Cancer 125, 2844–2853.
4. Fernandez-Zapico, M. E., Lomberk, G. A., Tsuji, S., DeMars, C. J., Bardsley, M. R., Lin, Y. H., et al. (2011). A functional family-wide screening of SP/KLF proteins identifies a subset of suppressors of KRAS-mediated cell growth. Biochem. J. 435, 529–537.
5. Ge, Y., Schuster, M. B., Pundhir, S., Rapin, N., Bagger, F. O., Sidiropoulos, N., et al. (2019). The splicing factor RBM25 controls MYC activity in acute myeloid leukemia. Nat. Commun. 10:172.
6. Zang, Y. S., Dai, C., Xu, X., Cai, X., Wang, G., Wei, J., et al. (2019). Comprehensive analysis of potential immunotherapy genomic biomarkers in 1000 Chinese patients with cancer. Cancer Med. 8, 4699–4708.
图片来源:领星