前言
T 细胞急性淋巴细胞白血病(T-ALL)是一种儿童中常见的恶性肿瘤,由于未成熟的 T 细胞克隆增殖导致。随着测序技术的不断发展,NGS 技术提供了与 T-ALL 发病机制相关的各种基因组损伤/病变的详细概述。研究发现,在 T-ALL 中,CDKN2A/2B 和 NOTCH1 是最容易受到影响的基因。此外,在诊断中发现 T-ALL 细胞中有 10 到 20 个蛋白质改变的病变。然而,目前还不清楚这些突变是以怎样的顺序获得的,以什么顺序启动了这些祖细胞。
为了解决以上问题,本研究对 4 例 T-ALL 患者的全骨髓细胞和 CD34+CD38-多能干细胞进行靶向单细胞测序(DNA 和 RNA 单细胞测序)。
结果分析
1、利用全基因组和转录组测序鉴定体细胞突变
对 4 个 T-ALL 儿童的骨髓样本进行全基因组和 RNA-seq,测序结果发现,每个病人的肿瘤样本平均有 10 个编码变异。此外,发现了一些融合基因、大的缺失和体细胞突变存在于 5'-UTR 和 3'-UTR 区以及剪切位点。

Fig. 1 Somatic variant identification by bulk sequencing of four primary T-ALL samples
2、靶向单细胞测序和质控
我们从每个病人中分离了 333 个白血病细胞。对单细胞基因组扩增后,利用病人特异性 primer 对感兴趣的区域进行测序。结合单细胞的突变型等位基因频率(VAF)比较每个突变的 VAFs,发现总体上呈良好的相关性,除了 XB47 患者,其单细胞 VAF 在所有突变中始终低于整体的 VAF。
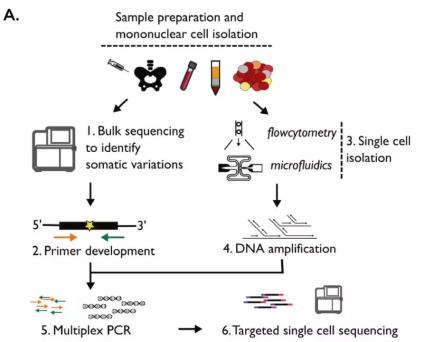
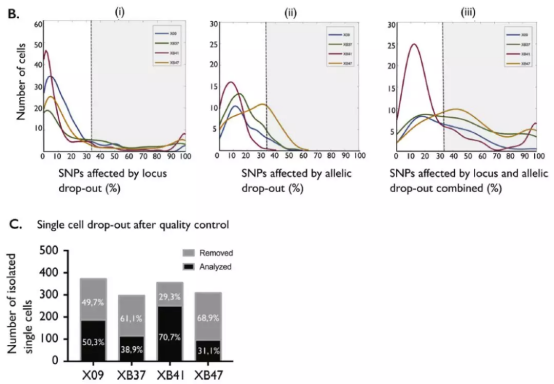
Fig. 2 Targeted single-cell sequencing of four primary T-ALL samples
为了过滤低质量细胞,我们研究了每个细胞杂合性 SNP 的位点和等位基因脱落(ADO)率。将位点丢失率(LDO)定义为 SNPS 与<4 reads 的比例。高 LDO 比例表明细胞中的许多 SNP 具有低覆盖率,表明基因组的较大区域未扩增。ADO 表明,如果只有一个等位基因被扩增,SNP 的杂合度可能会丢失。之前的研究表明,质控之后只有那些 SNP 中少于 1/3 受 LDO 和 ADO 联合影响的细胞被认为具有足够的质量并用于进一步分析。在 1332 个分离的单白血病细胞中,649 个符合这些标准,且这些细胞数量在不同患者之间有显著性差异。
3、靶向单细胞测序揭示了 4 个 T-ALL clusters
对测序数据进行聚类分析,发现每个病人都有一个高度突变的优势细胞群体,从 28-94% 不等,并伴随着一些较少突变的小簇。
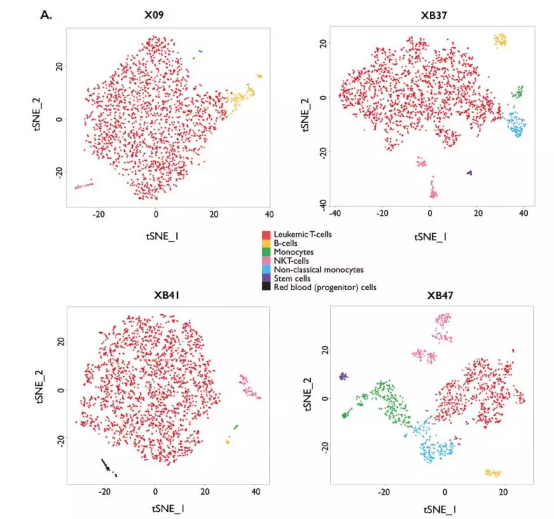
Fig. 3 T-ALL patient samples have limited heterogeneity at presentation. Columns represent single cells, rows represent the somatic variations.
4、单细胞测序揭示了 T-ALL 细胞的转录均匀性
平均每个病人分析 2,074 个细胞,以进一步探究 T-ALL 样本的异质性。结果发现白血病细胞始终聚类在一起,表明在 T-ALL 细胞之间的转录异质性有限。其他簇代表正常 B 细胞、单核细胞、NK T 细胞、干细胞,甚至少数产生血红蛋白的红细胞 (祖细胞)。
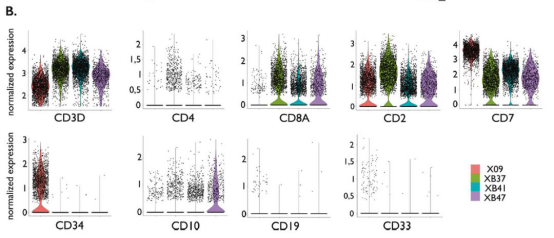
CD19 and CD33 expression represent negative controls
对白血病细胞的一些表面 marker 的基因表达进行评估,并与其诊断时的免疫表型进行匹配。与靶向 DNA 测序数据一致,XB47 患者只含有 47% 的白血病 T 细胞。
5、T-ALL 突变可以在 CD34+ CD38- 多能干细胞中启动
CD34+ CD38- 细胞与其他样本一样的测序和质控标准。
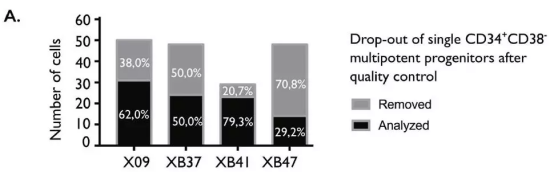
4 个 T-ALL 患者中有 2 个患者(X09, XB41)的 CD34+CD38-祖细胞显示高度突变的特征。大多数这些突变也在诊断性骨髓定向祖细胞中检测到,与患者在干细胞或多能祖细胞中获得的突变相一致。但是,患者的病情缓解后,这些高度突变的多能祖细胞就可以被清除掉。相反,对于另外 2 个病人(XB37, XB47),在 CD34+CD38-细胞和骨髓祖细胞中检测到的突变非常少,表明这些突变中的大多数是在已经致力于淋巴系祖细胞中获得的。
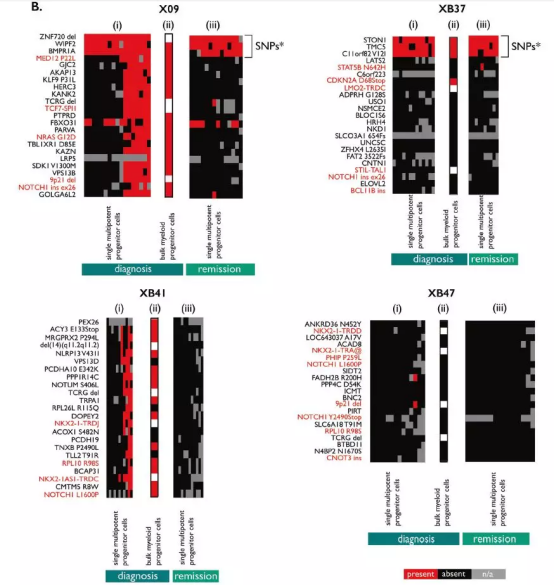
Fig. 5 Multiple mutations can be present in multipotent progenitor cells.
6、靶向单细胞测序可以确定 T-ALL 中获得突变的顺序
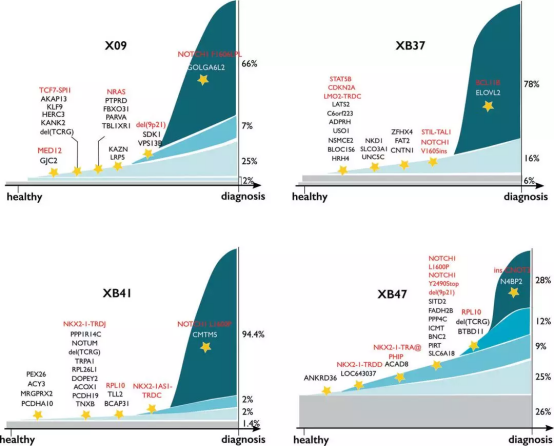
Fig. 6 Single-cell data illuminate the mutational hierarchy in T-ALL patient samples.